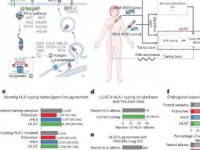
多伦多大学研究人员使用生成式AI设计新型蛋白质
多伦多大学的研究人员 开发了一种人工智能系统,可以使用生成扩散创建自然界中不存在的蛋白质,这与 DALL-E 和 Midjourney等流行图像创建平台背后的技术相同。

该系统将有助于推进生成生物学领域的发展,该领域有望通过使全新治疗性蛋白质的设计和测试更加高效和灵活来加速药物开发。
“我们的模型从图像表示中学习,以非常高的速度生成全新的蛋白质,” 多伦多大学 Temerty 医学院唐纳利细胞和生物分子研究中心教授 Philip M. Kim说。“我们所有的蛋白质在生物物理学上似乎都是真实的,这意味着它们折叠成使它们能够在细胞内执行特定功能的配置。”
今天, Nature Computational Science 杂志 发表了 这一发现,这是同行评议期刊中的首例此类发现。去年夏天, Kim 的实验室还 通过开放访问服务器 bioRxiv 发布了该模型的 预印本,领先于去年 12 月的两份类似预印本, 即华盛顿大学的 RF Diffusion和 Generate Biomedicines 的Chroma 。
蛋白质由折叠成三维形状的氨基酸链制成,这反过来又决定了蛋白质的功能。这些形状经过数十亿年的演变,变化多样且复杂,但数量有限。随着对现有蛋白质折叠方式的更好理解,研究人员开始设计非自然产生的折叠模式。
但 Kim 说,一个主要的挑战是想象既可行又实用的折叠。“很难预测哪些折叠是真实的并在蛋白质结构中发挥作用,”金说,他也是多伦多大学分子遗传学和计算机科学系的教授。“通过结合基于 生物物理学 的 表征通过图像生成空间的扩散方法来构建蛋白质结构,我们可以开始解决这个问题。”
研究人员称之为 ProteinSGM 的新系统从大量现有蛋白质的类图像表示中提取,这些蛋白质准确地编码了它们的结构。研究人员将这些图像输入一个生成扩散模型,该模型逐渐添加噪声,直到每张图像都变成噪声。该模型跟踪图像如何变得更嘈杂,然后反向运行该过程,学习如何将随机像素转换为与全新蛋白质相对应的清晰图像。
Kim 实验室的博士生、该论文的第一作者Jin Sub (Michael) Lee表示,优化图像生成过程的早期阶段是创建 ProteinSGM 的最大挑战之一。“一个关键的想法是蛋白质结构的适当图像式表示,这样扩散模型就可以学习如何准确地生成新蛋白质,”来自温哥华但之前在韩国获得本科学位并在瑞士获得硕士学位的李说。选择多伦多大学攻读博士学位。
同样困难的是验证 ProteinSGM 生产的蛋白质。该系统生成许多结构,通常不同于自然界中发现的任何结构。Lee 说,根据标准指标,几乎所有这些看起来都是真实的,但研究人员需要进一步的证据。
为了测试他们的新蛋白质,Lee 和他的同事首先求助于 OmegaFold,它是 DeepMind 软件 AlphaFold 2的改进版本。这两个平台都使用 AI 根据氨基酸序列预测蛋白质的结构。
通过 OmegaFold,该团队证实几乎所有新序列都折叠成所需的新蛋白质结构。然后他们选择了一个较小的数字在试管中进行物理创建,以确认这些结构是蛋白质,而不仅仅是杂散的化合物串。
“通过 OmegaFold 中的匹配和实验室中的实验测试,我们可以确信这些是正确折叠的蛋白质。看到这些在自然界中任何地方都不存在的全新蛋白质折叠得到验证,真是太神奇了,”Lee 说。
Kim 说,基于这项工作的下一步包括进一步开发用于抗体和其他具有最大治疗潜力的蛋白质的 ProteinSGM。“对于研究和创业来说,这将是一个非常令人兴奋的领域,”他补充道。
Lee 说他希望看到生成生物学朝着蛋白质序列和结构的联合设计方向发展,包括蛋白质侧链构象。迄今为止,大多数研究都集中在骨干的生成上,骨干是将蛋白质结合在一起的主要化学结构。
“侧链配置最终决定了蛋白质的功能,虽然设计它们意味着复杂性呈指数级增加,但通过适当的工程设计可能是有可能的,”Lee 说。“我们希望找出答案。”
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。