检测蛋白质折叠过程中的新状态
科学家们在蛋白质折叠过程中发现了一种新的中间状态,表明折叠可以分两个阶段发生,一个阶段很快,而下一个阶段则慢得多。研究结果发表在《物理评论快报》杂志上。
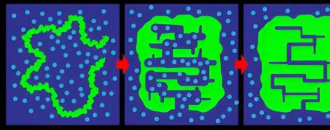
蛋白质(由多达20个不同氨基酸分子组成的链)在细胞中产生后,它会自发地从随机结构卷曲(“折叠”)成有序的三维结构,从而使其具有生物学用途。(折叠不当可能导致肌萎缩侧索硬化症(ALS,也称为卢伽雷氏病)等疾病。)
对于更简单的蛋白质,这通常发生得非常快,大约需要0.01毫秒(ms),直到其接近完整的形式。在这项研究中,由于蛋白质侧链的小幅重排,在第一步之后发现出现了第二个较慢的状态,持续时间为3-10毫秒,比之前的折叠时间长了一千倍。(相比之下,人眼的眨眼持续时间在100到400毫秒之间。)
科学家们已经学会利用机器学习来预测蛋白质将折叠成什么样的结构。但人们对为什么每种蛋白质会产生不同的特定结构,以及当各种氨基酸彼此发生化学反应时如何发生这一现象,人们知之甚少。
先前的分析方法通过光谱(测量左旋和右旋圆偏振光吸收方式之间的差异)等方法来观察蛋白质在旋转和扭曲成卷曲、折叠时如何从其内部挤出水分。状态。然而,这种光学测量并不能捕捉到这里发现的最终的长期折叠。
美国国立卫生研究院国家糖尿病、消化和肾脏疾病研究所(NIDDK)化学物理实验室代理主任罗伯特·泰科(RobertTycko)领导的研究人员决定寻找这种长期折叠。
他们选择了以简单和快速折叠而闻名的蛋白质HP35,由35个氨基酸残基组成。(残基是两个氨基酸分子之间连接的结果,形成肽,从而去除水。这些残基影响其他残基以及整个蛋白质的化学性质。)也称为“villinheadpiecesubdomain”,折叠研究中常用的蛋白质。
该小组利用热量来展开HP35,在25毫秒内将其加热至95°C。然后,他们让它在30°C的温暖条件下,在约0.1毫秒的“孵化期”内重新折叠。他们将蛋白质在该温度下保温1至10毫秒,然后将其快速冷冻至-35°C,此时形成直径约0.3毫米的颗粒。
使用光学光谱探针观察HP35的快速折叠后,使用蛋白质残基中碳13原子的固态核磁共振分析冷冻颗粒的浆液,以观察它们在初始快速折叠后的行为。在13C的核自旋特征振荡频率下,当分子结构变得更加刚性时,振荡施加磁场的信号变得更加尖锐。
研究小组观察到,两个特定残基的13C共振频率在孵育期间没有变化,表明它们是在孵育期开始时已经折叠的蛋白质部分。然而,来自蛋白质其他部分的几个其他残基的共振频率的信号在10毫秒的孵育期内变得尖锐,表明这些蛋白质部分在孵育期开始后仍在折叠。
也就是说,HP35在潜伏期开始时似乎在0.1毫秒内折叠,但实际上只有在10毫秒潜伏期结束后才完全折叠,最终形式变得刚性。
这一结果是折叠过程中形成所谓的DMG(干熔球)状态的第一个明确证据。DMG于1989年首次在理论上被提出为折叠蛋白所采取的倒数第二个状态,在此之前,仅观察到湿熔球状态,其中预先形成的蛋白质在水中处于更大的构型。
到目前为止,还没有DMG状态的证据,但有人认为在研究各种蛋白质的展开过程中它是存在的。另一项对酶(加速化学反应的催化剂蛋白)的研究提供了折叠可能持续数天的暗示。
HIDDK的Tycko表示:“结构优化或退火的第二阶段在之前的研究中并未观察到,但在我们的实验中却出现了,因为我们测量了NMR信号,这些信号对构象变化和局部结构细节非常敏感。”
他指出,许多测量亚毫秒温度跃变的设备都是在他的实验室开发的,他补充说,“利用这些设备,我们可以在涉及大的单向结构变化的其他生物分子过程中寻找类似的效应。”
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。