拟议的新指标用于量化个体与训练群体之间的遗传差异
隶属于美国多个机构和丹麦奥胡斯大学的一组生物信息学研究人员提出了一种新指标,用于量化个体与训练人群之间的遗传差异。他们的研究报告在《自然》杂志上。《自然》杂志的编辑还在同一期期刊上发表了一份研究简报,概述了该团队在这方面所做的工作。
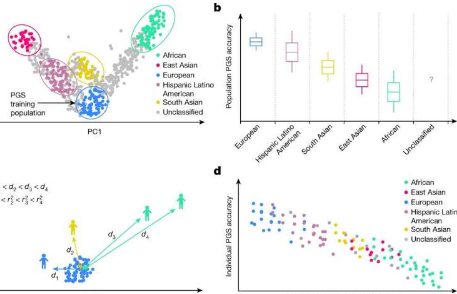
多基因评分(PGS)是一种工具,用于根据遗传背景估计特定性状或疾病的概率。PGS通常是通过将许多与感兴趣的性状相关的常见遗传变异的影响相加来计算的。但衍生分数的准确性取决于用于构建它们的遗传变异在多大程度上实际捕获了它们所来自的种群的遗传多样性。
这通常意味着,如果用于训练PGS的给定人群在遗传上与应用测试的人群不同,则PGS可能表现不佳。为了使这些分数更有用,研究人员提出了一种称为遗传距离(GD)的新指标——其目的是根据全基因组等位基因频率量化个体与训练群体之间的遗传差异。
新指标的范围从0(代表相同的特征)到1(代表完全不同的特征)——它还将考虑影响特定人群的古代和近期进化事件。为了支持新指标的使用,研究小组表明,对于某些疾病和跨人群的特征,即使那些通常被认为是同质的,GD也可能与PGS呈负相关。该团队还证明,GD可用于识别可能受益于针对特定人群训练的PGS的人,或者相反,那些更多样化的人——或依赖于不同变体集的PGS。
该团队得出结论,他们的指标可以为衡量PGS的准确性提供连续的衡量标准,并指出它还强调了在开发PGS时考虑遗传多样性的重要性。
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。