研究人员建立发酵原核生物的交互式综合数据库
大多数生物利用氧气将食物转化为能量。然而,在氧气很少或没有氧气的环境中,生命找到了其他产生能量的方法,即使用一种称为发酵的过程。
发酵代谢发生在反刍动物肠道、沉积物和厌氧生物反应器等环境中。除了产生生物体可以用来繁衍的能量外,该过程还可以产生其他最终产品,包括生物燃料和其他生物产品。
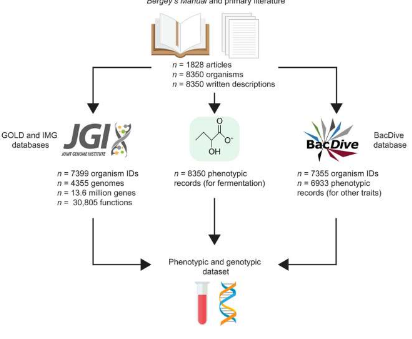
为了更好地了解依赖这种新陈代谢形式的细菌和古细菌的范围,研究人员最近编制了一份清单,其中包含来自多个来源的 8,300 多种生物体,其中包括美国能源部 (DOE) 联合基因组研究所 (JGI)科学用户设施办公室位于劳伦斯伯克利国家实验室(伯克利实验室)和能源部系统生物学知识库(KBase)。
他们还构建了一个交互式浏览器,允许其他研究人员研究基因组并预测可能在缺氧或无氧环境中繁衍生息的微生物的代谢能力。该研究发表在《科学进展》上。
加州大学戴维斯分校和加州大学圣巴巴拉分校的研究人员从文献中生成了具有发酵代谢功能的原核生物列表。然后,他们使用 JGI 的 GOLD 和 IMG 数据门户来获取有关列表中生物体的基因组和基因的信息。他们还在 IMG/M 中搜索了属于每个基因组的所有蛋白质编码基因。
最终数据集(研究界可以通过在线交互式浏览器获取)包含 8,350 个生物体的表型记录以及 4,355 个基因组和 1,360 万个基因。结果表明,三分之一的原核生物存在发酵代谢,可形成大约 300 种代谢物组合。
为了演示该浏览器的潜在实用性,该团队使用 Fermentation Explorer 和一系列大型数据集。一项测试是预测 Hungate1000 项目中 400 多种培养的瘤胃原核生物的性状。他们还使用该工具预测 733 个宏基因组组装的瘤胃基因组的可能功能。最后,他们使用该工具预测了五种以前未表征的原核生物的发酵最终产物,其中包括他们实验室分离的两种细菌。
KBase 用于快速组装和注释这两个分离株的分类和功能。组合的分类学和功能注释用于预测这些基因组的最终产物。然后,该团队培养了这些生物体,发现预测的最终产品与实际的最终产品基本一致。
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。