完善微生物群落样本的生物群落标记
在《环境科学与生态技术》杂志上发表的一项研究中,华中科技大学的研究人员推出了“Meta-Sorter”,这是一种基于人工智能的方法,利用神经网络和迁移学习来显着改善数千个微生物组样本的生物组标记MGnify数据库中,特别是那些信息不完整的。
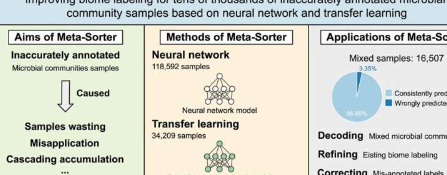
元排序方法包括两个关键步骤。首先,使用来自134个生物群落的118,592个微生物样本及其各自的生物群落本体精心构建了神经网络模型,其平均AUROC高达0.896,令人印象深刻。该模型利用详细的生物群落信息对样本进行准确分类,为进一步分析奠定了坚实的基础。
其次,为了应对新引入的具有不同特征的样本的挑战,研究人员将迁移学习与来自35个生物群落的34,209个新添加的样本结合起来,其中包括8个新样本。转移神经网络模型取得了0.989的出色平均AUROC,成功预测了新引入的标注为“混合生物群落”的样本的生物群落信息。
Meta-Sorter的结果确实令人印象深刻,在16,507个缺乏详细生物群落注释的样本中实现了96.7%的总体准确率。这一突破有效地解决了级联错误的问题,并为各个科学学科(特别是环境研究)的知识发现开辟了令人兴奋的新可能性。
此外,Meta-Sorter的成功还延伸到改进了注释不足和错误注释样本的生物群落注释。它对模糊样本进行智能、自动的精确分类,提供了超出原始文献的宝贵见解,而将样本区分为特定环境类别则增强了研究结论的可靠性和有效性。
随着数据提交和纳入附加元数据信息的标准化协议的不断发展,Meta-Sorter将彻底改变研究人员分析和解释微生物群落样本的方式。最终,它将在微生物组研究及其他领域带来更准确和更有洞察力的发现。
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。










